Nota
Fare clic qui per scaricare il codice di esempio completo
Normalizzazioni della mappa dei colori #
Dimostrazione dell'uso della norma per mappare le mappe dei colori sui dati in modi non lineari.
import numpy as np
import matplotlib.pyplot as plt
import matplotlib.colors as colors
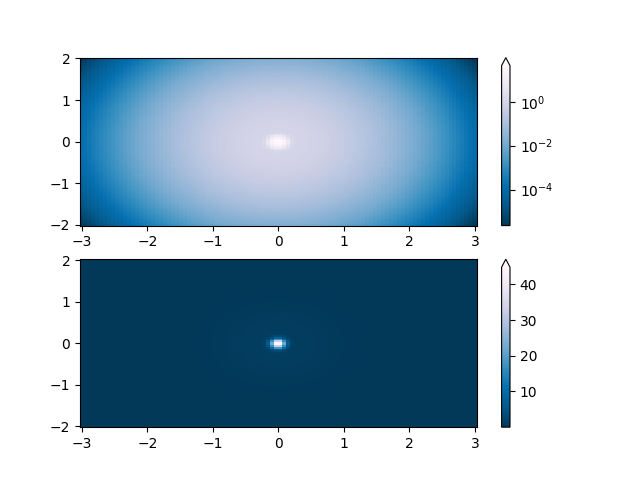
Lognorm: invece di pcolor log10(Z1) puoi avere barre dei colori che hanno le etichette esponenziali usando una norma.
N = 100
X, Y = np.mgrid[-3:3:complex(0, N), -2:2:complex(0, N)]
# A low hump with a spike coming out of the top. Needs to have
# z/colour axis on a log scale so we see both hump and spike. linear
# scale only shows the spike.
Z1 = np.exp(-X**2 - Y**2)
Z2 = np.exp(-(X * 10)**2 - (Y * 10)**2)
Z = Z1 + 50 * Z2
fig, ax = plt.subplots(2, 1)
pcm = ax[0].pcolor(X, Y, Z,
norm=colors.LogNorm(vmin=Z.min(), vmax=Z.max()),
cmap='PuBu_r', shading='nearest')
fig.colorbar(pcm, ax=ax[0], extend='max')
pcm = ax[1].pcolor(X, Y, Z, cmap='PuBu_r', shading='nearest')
fig.colorbar(pcm, ax=ax[1], extend='max')
<matplotlib.colorbar.Colorbar object at 0x7f2cfb030d90>
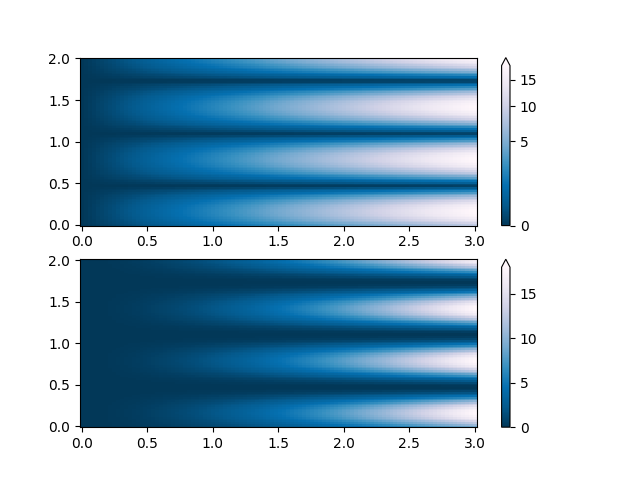
PowerNorm: qui un andamento della legge di potenza in X oscura parzialmente un'onda sinusoidale rettificata in Y. Possiamo rimuovere la legge di potenza usando una PowerNorm.
X, Y = np.mgrid[0:3:complex(0, N), 0:2:complex(0, N)]
Z1 = (1 + np.sin(Y * 10.)) * X**2
fig, ax = plt.subplots(2, 1)
pcm = ax[0].pcolormesh(X, Y, Z1, norm=colors.PowerNorm(gamma=1. / 2.),
cmap='PuBu_r', shading='nearest')
fig.colorbar(pcm, ax=ax[0], extend='max')
pcm = ax[1].pcolormesh(X, Y, Z1, cmap='PuBu_r', shading='nearest')
fig.colorbar(pcm, ax=ax[1], extend='max')
<matplotlib.colorbar.Colorbar object at 0x7f2cfb4f44c0>
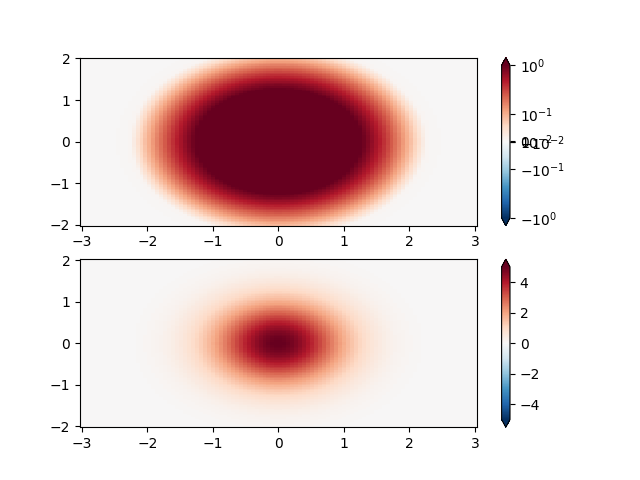
SymLogNorm: due gobbe, una negativa e una positiva, la positiva con 5 volte l'ampiezza. Linearmente, non puoi vedere i dettagli nella gobba negativa. Qui ridimensioniamo logaritmicamente i dati positivi e negativi separatamente.
Si noti che le etichette della barra dei colori non hanno un bell'aspetto.
X, Y = np.mgrid[-3:3:complex(0, N), -2:2:complex(0, N)]
Z1 = 5 * np.exp(-X**2 - Y**2)
Z2 = np.exp(-(X - 1)**2 - (Y - 1)**2)
Z = (Z1 - Z2) * 2
fig, ax = plt.subplots(2, 1)
pcm = ax[0].pcolormesh(X, Y, Z1,
norm=colors.SymLogNorm(linthresh=0.03, linscale=0.03,
vmin=-1.0, vmax=1.0, base=10),
cmap='RdBu_r', shading='nearest')
fig.colorbar(pcm, ax=ax[0], extend='both')
pcm = ax[1].pcolormesh(X, Y, Z1, cmap='RdBu_r', vmin=-np.max(Z1),
shading='nearest')
fig.colorbar(pcm, ax=ax[1], extend='both')
<matplotlib.colorbar.Colorbar object at 0x7f2cfb59c9a0>
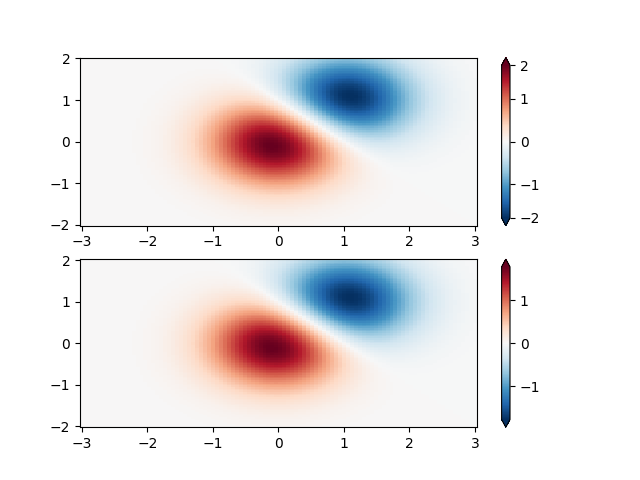
Norma personalizzata: un esempio con una normalizzazione personalizzata. Questo utilizza l'esempio precedente e normalizza i dati negativi in modo diverso da quelli positivi.
X, Y = np.mgrid[-3:3:complex(0, N), -2:2:complex(0, N)]
Z1 = np.exp(-X**2 - Y**2)
Z2 = np.exp(-(X - 1)**2 - (Y - 1)**2)
Z = (Z1 - Z2) * 2
# Example of making your own norm. Also see matplotlib.colors.
# From Joe Kington: This one gives two different linear ramps:
class MidpointNormalize(colors.Normalize):
def __init__(self, vmin=None, vmax=None, midpoint=None, clip=False):
self.midpoint = midpoint
super().__init__(vmin, vmax, clip)
def __call__(self, value, clip=None):
# I'm ignoring masked values and all kinds of edge cases to make a
# simple example...
x, y = [self.vmin, self.midpoint, self.vmax], [0, 0.5, 1]
return np.ma.masked_array(np.interp(value, x, y))
#####
fig, ax = plt.subplots(2, 1)
pcm = ax[0].pcolormesh(X, Y, Z,
norm=MidpointNormalize(midpoint=0.),
cmap='RdBu_r', shading='nearest')
fig.colorbar(pcm, ax=ax[0], extend='both')
pcm = ax[1].pcolormesh(X, Y, Z, cmap='RdBu_r', vmin=-np.max(Z),
shading='nearest')
fig.colorbar(pcm, ax=ax[1], extend='both')
<matplotlib.colorbar.Colorbar object at 0x7f2cfb533910>
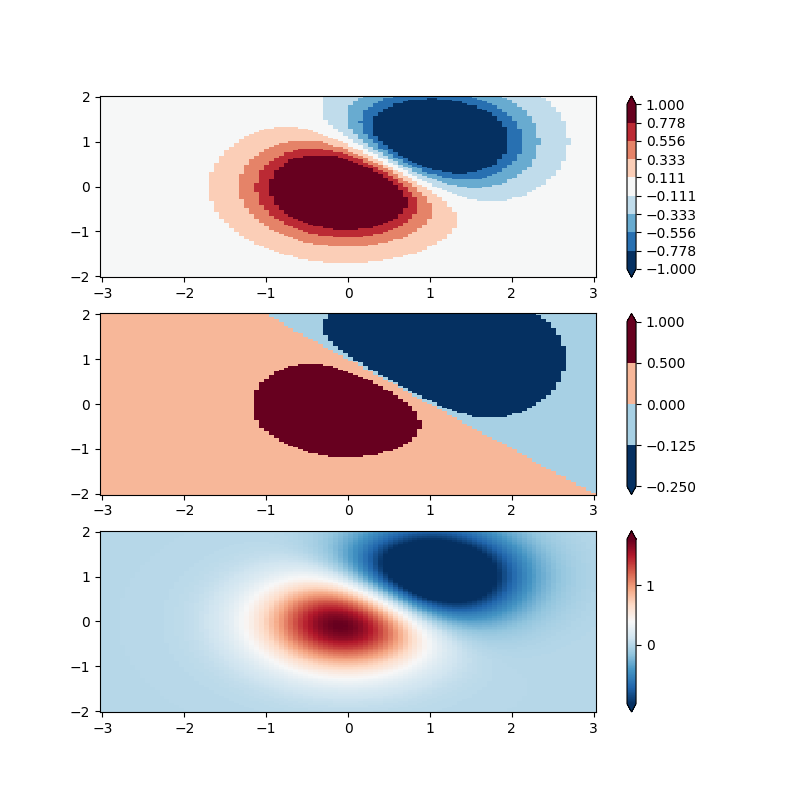
BoundaryNorm: per questo fornisci i confini per i tuoi colori, e la norma mette il primo colore tra la prima coppia, il secondo colore tra la seconda coppia, ecc.
fig, ax = plt.subplots(3, 1, figsize=(8, 8))
ax = ax.flatten()
# even bounds gives a contour-like effect
bounds = np.linspace(-1, 1, 10)
norm = colors.BoundaryNorm(boundaries=bounds, ncolors=256)
pcm = ax[0].pcolormesh(X, Y, Z,
norm=norm,
cmap='RdBu_r', shading='nearest')
fig.colorbar(pcm, ax=ax[0], extend='both', orientation='vertical')
# uneven bounds changes the colormapping:
bounds = np.array([-0.25, -0.125, 0, 0.5, 1])
norm = colors.BoundaryNorm(boundaries=bounds, ncolors=256)
pcm = ax[1].pcolormesh(X, Y, Z, norm=norm, cmap='RdBu_r', shading='nearest')
fig.colorbar(pcm, ax=ax[1], extend='both', orientation='vertical')
pcm = ax[2].pcolormesh(X, Y, Z, cmap='RdBu_r', vmin=-np.max(Z1),
shading='nearest')
fig.colorbar(pcm, ax=ax[2], extend='both', orientation='vertical')
plt.show()
Tempo di esecuzione totale dello script: (0 minuti 3,715 secondi)